Высокое генетическое разнообразие и обнаружение нового подтипа HCV-6 среди иммигрантов азиатского происхождения в Северной Америке
Введение
Вирус гепатита С (HCV) ежегодно фиксируется примерно у 115 миллионов человек во всем мире (Gower et al., 2014). Хроническая инфекция HCV представляет собой одну из основных причин развития цирроза печени и гепатоцеллюлярной карциномы (WHO, 2014). HCV классифицируется на семь генотипов, которые имеют разницу в нуклиотидной последовательности примерно около 30% (Smith et al., 2014, Murphy et al., 2015). Генотипы подразделяются на подтипы, и в настоящее время выявлено, установлено и общепризнано более 87 подтипов. Подтипы обычно различаются на ≥15% (Simmonds et al., 2005, Smith et al., 2014). Генотип HCV является важным фактором получения устойчивого вирусологического ответа как на комбинированную терапию пэгилированным интерфероном и рибавирином у пациентов с хроническим поражением (EASL, 2011 г.). Использование противовирусных препаратов прямого действия (DAAs) и их эффективность также тесна связана с генотипом HCV, в свою очередь, частота резистентности, по-видимому, варьируется в зависимости от гетерогенности целевых вирусных белков, таких как NS3/4A (Ippolito et al., 2014, Soriano et al., 2011). Распределение и распространенность генотипов HCV коррелируют с характером передачи вируса и миграции в популяции (Pybus et al., 2009, Lu et al., 2014a). По этой причине характерные особенности связанные с конкретным генотипом HCV имеют как клиническую, так и эпидемиологическую значимость.
Среди семи генотипов HCV именно шестой (HCV-6) проявляет наибольшее генетическое разнообразие, и по устоявшемуся мнению, считается, что он представляет собой самую старую линию HCV (Salemi and Vandamme, 2002, Wang et al., 2013). Основываясь на последних исследованиях которые провела наша группа, HCV-6 на сегодня классифицируется по 28 подтипам 6a-6xe, каждый из которых имеет по крайней мере одну известную полноразмерную последовательность генома. В настоящем исследовании мы описали полную последовательность геномов, каждая из которых, вероятно, представляет новый подтип. Это увеличило количество полностью упорядоченных неклассифицированных таксономических линий HCV-6 до 34.
Географически изоляты HCV-6 были в основном обнаружены в Юго-Восточной Азии или среди иммигрантов в Северной Америке прибывших из этого региона, что может указывать на потенциальную область происхождения (Bernier et al., 1996, Mellor et al., 1996, Noppornpanth et al., 2006, Pham et al., 2011, Pybus et al., 2009, Shinji et al., 2004, Stuyver et al., 1995, Thaikruea et al., 2004, Theamboonlers et al., 2002). Они включали широкий спектр изолятов HCV-6, обнаруженных в Квебеке, Канада (Murphy et al., 2007) и более низкое число в США (Lu et al., 2007b). Было установлено что на территории США и Канады HCV-6 составляет приблизительно 1,8% от общего количества пациентов с HCV-инфекцией (Nainan et al., 2006, Murphy et al., 2007), при этом, почти все изоляты HCV-6 были отобраны у азиатских иммигрантов. В текущем исследовании мы охарактеризовали 20 изолятов HCV-6: 16 из Канады, 4 из США. Знание данных генетической информации значительно улучшат наши знания о разнообразии HCV-6 в Северной Америке, что может способствовать будущим исследованиям по профилактике и борьбе с HCV-инфекцией на континенте.
Геномные последовательности
Почти полноразмерный геном HCV-6 характеризовался 15 изолятами: 745291, QC81, QC112, QC150, QC197, QC251, QC255, QC266, QC269, QC287, QC176, QC189, QC120, QC292 и QC334, каждый имел 16 перекрывающихся фрагментов. Эти геномы имели длину 9409-9539 нуклеотидов (nt), начиная с крайней 5′-нераздраженной области (UTR) до переменной области или X-хвоста 3′-UTR. Каждый из них содержал одну открытую рамку считывания 9039-9114 н.т. 5′-UTR составляли 338-339 нт, а 3′-UTR варьировались от 22 до 153 нт. Размеры десяти областей кодирующих белок были такими же, как те, что указаны в геноме H77, за исключением областей E2 (1083-1101 н. Т) и NS5A (1053-1101 aa), которые имели изменчивость среди 15 изолятов, а также в области NS5B QC112. Последний имел вставку 54 нт (18 аа) между nt, пронумерованными 8327 и 8328 в геноме H77.
Анализ последовательностей ORF
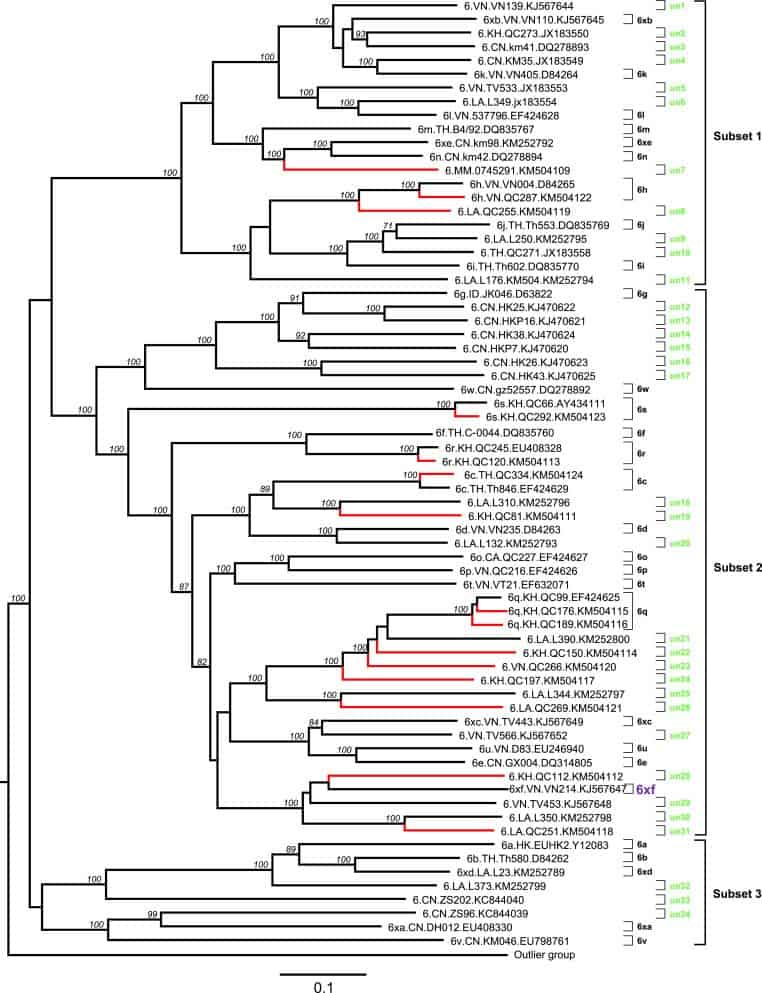
Филогенция максимального правдоподобия (ML) была восстановлена на основе 69 последовательностей HCV-6 по всей длине ORF вместе с одной контрольной последовательностью от каждого из шести других генотипов (рис.1). Последние шесть были спрессованы в одну ветвь, чтобы сформировать группу (рис.1). 69 последовательностей HCV-6 были разделены на 29 подтипов, 6a-6xf и 34 неклассифицированных линиях. Из 15 геномов, определенных в этом исследовании, QC334 был классифицирован в подтип 6c (p-расстояние от Th846 = 9,0%), QC287 — подтип 6h (p-расстояние от VN004 = 8,9%), QC292 — подтип 6s (p-расстояние от QC66 = 5,8%), QC120 в подтип 6r (p-расстояние от QC245 = 4,1%) и QC176 и QC189 в подтип 6q (p-расстояния от QC99 = 5,6-5,9%). Основываясь на критериях текущей классификации ВГС (Simmonds et al., 2005, Smith et al., 2014), такие различия были в пределах подтипа. Поэтому мы классифицировали эти шесть геномов в описанные пять подтипов.
Оставшиеся девять геномов, охарактеризованные в этом исследовании, были сгруппированы за пределами 29 подтипов, 6a-6xf. Для удобства описания мы разделили кластер HCV-6 на три подмножества (рис.1). С вершины дерева подмножество 1 содержало два (0745291 и QC255) из девяти геномов, упомянутых выше, подмножество 2 включало семь (QC81, QC150, QC266, QC197, QC269, QC112 и QC251), тогда как подмножество 3 не содержало ни одного. В подмножестве 1 0745291 наиболее тесно связано с подтипами 6m (B4-92), 6n (KM42) и 6xe (KM98), имеющим p-расстояния 20,1%, 19,2% и 19,3% соответственно. QC255 был наиболее тесно связан с подтипом 6h (D84265 и QC287), имея p-расстояния 15,4%. В подмножестве 2 QC81 был наиболее тесно связан с неклассифицированной последовательностью L310 (p-distance = 16,9%). QC150, QC197 и QC266 были наиболее тесно связаны друг с другом (p-расстояния = 18,3-18,8%) и с неклассифицированной последовательностью L390 (p-расстояния = 18,2%, 18,5% и 17,8%). QC269 наиболее тесно связан с неклассифицированной последовательностью L344 (p-distance = 20,6%), а QC251 наиболее тесно связан с неклассифицированной последовательностью L350 (p-расстояние = 15%). В совокупности эти девять геномов показали, что p-расстояния были в пределах ≥15% от ближайших родственных линий.
Рисунок 1. Дерево на основе 75 полноразмерных последовательностей генома HCV. 15 геномов, охарактеризованных в этом исследовании, указаны в красных ветвях. Они были проанализированы вместе с 54 дополнительными последовательностями полной длины HCV-6 и сравнены с шестью другими генотипами. Для упрощения последние шесть сжимаются в одну ветвь и показаны на древовидной базе. Все подсказки указаны вформате: генотип или подтип. Идентификация страны. Идентификационный номер ID.GenBank.
Изоляты HCV-6 в Северной Америке
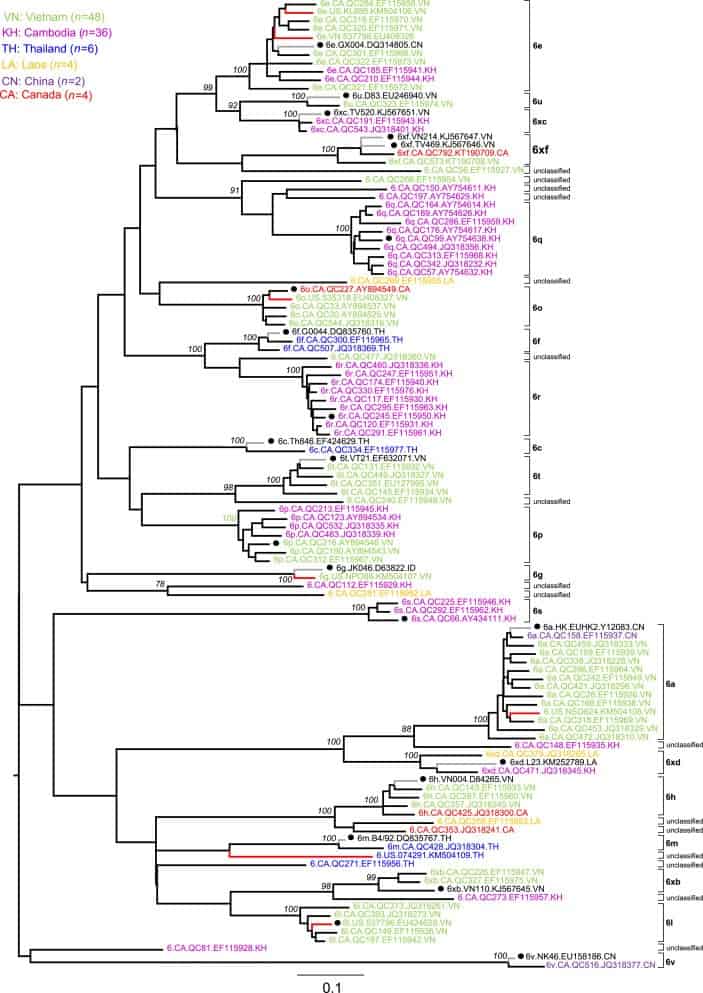
Чтобы продемонстрировать наличие в Северной Америке разнообразных штаммом HCV-6, мы дополнительно проанализировали 115 изолятов HCV-6 в области Core-E1 (рис.2). Они включали 15 изолятов, описанных выше, пять дополнительных изолятов (KL885, NPO88, NSO624, QC573 и QC792), которые мы частично секвенировали в настоящем исследовании, 80 изолятов, о которых сообщалось ранее (Murphy et al., 2007, Lu et al. ., 2007b) и 15 опорных последовательностей подтипов HCV-6, которые были получены из GenBank (Smith et al., 2014). Исключая последние 15, остальные 100 последовательностей HCV-6 были отобраны в Северной Америке: семь в США и 93 в Канаде. Из этих 100 последовательностей 96 были получены от азиатских иммигрантов, а четверо от иммигрантов кавказского региона. Данные 100 последовательностей можно разделить на 20 условных подтипов и 16 неклассифицированных линий, демонстрирующих широкое генетическое разнообразие. Они включали 13 изолятов, 6 подтипов, десять 6е, девять из 6q и 6r, семь 6p, пять из 6l и 6o, четыре из 6h и 6t, три 6s, два из 6f, 6xb, 6xc, 6xd и 6xf , и один из 6c, 6g, 6m, 6u и 6v и 16 неклассифицированных вариантов. Среди 96 иммигрантов прибывших из стран Азии, у которых был обнаружен HCV-6, 48 родились во Вьетнаме, 36 в Камбодже, шесть в Таиланде, четыре в Лаосе и два в Китае (рис.2).
Подтип 6xf
Подтип 6xf был впервые обнаружен в данном исследовании. Недавно мы описали полноразмерные геномы для двух вариантов HCV-6 TV469 (KJ567646) и VN214 (KJ567647) с использованием образцов из Вьетнама (Li et al., 2014a). Филогенетический анализ показал, что эти два будут представлять новый подтип HCV-6, поскольку они показали >15% нуклеотидных различий по сравнению с другими известными последовательностями HCV-6. В связи с отсутствием идентификации трех тесно связанных, но несвязанных изолятов, они были классифицированы как неклассифицированная линия HCV-6. В этом исследовании частичные последовательности в областях Core-E1 и NS5B были получены для двух других близко родственных изолятов: QC573 (KT190708 и KT190710) и QC792 (KT190709 и KT190711), один от иммигранта из Вьетнама, а другой от иммигранта из кавказского региона. Параллельное сравнение последовательностей показало, что у четырех человек отличии составляли 7,9-14,2% нуклеотидов в области кодирующей Core-E1 и 5,6-8,3% в области кодирующей NS5B. Все эти различия попали в диапазон подтипов и позволили нам определить их новому подтипу 6xf (рис.2). Поскольку полноразмерные геномы были также определены как для TV469, так и для VN214, это наши данные можно подтвердить на основе текущих рекомендуемых критериев классификации HCV (Simmonds et al., 2005, Smith et al., 2014).
Рисунок 2. Дерево на основе данных 115 последовательностей core-E1 HCV-6. Черные круги обозначают последовательности условных подтипов (Smith et al., 2014). Красные ветви представляют последовательности, отобранные в США. Серые ветви представляют собой эталонные последовательности из других стран. Остальные последовательности были отобраны в Канаде.
Обсуждение
В этом исследовании шесть полноразмерных геномов были разделены на подтипы 6c, 6h, 6q, 6r и 6s. Для подтипов 6c и 6q описанные последовательности представляют собой второй полноразмерный геном и служат для проверки подтипов. Мы также достоверно установили что существует новый подтип — 6xf (Simmonds et al., 2005, Smith et al., 2014); это определение значительно расширяет таксономическую структуру HCV-6, которая теперь включает в себя в общей сложности 29 подтипов.
Девять полноразмерных геномов из этого исследования не группировались в 29 определенных подтипах 6a-6xf. В одном из наших недавних исследований анализ частичных NS5B-последовательностей из этих девяти изолятов показал, что они не смогли быть сгруппированны с любыми другими штаммами HCV-6 и, таким образом, представляют собой уникальные варианты (Li et al., 2014a, Li et al., 2015). Это открытие подтверждено в данном исследовании на основе филогенетического анализа их полных последовательностей генома. Обнаружено, что они расходятся друг от друга и от ближайших родственников на ≥15%. Это нижний предел нуклеотидных различий, показанный для выделения изолятов разных подтипов (Simmonds et al., 2005, Smith et al., 2014). Однако, поскольку каждый из них не имеет трех тесно связанных изолятов, они по-прежнему классифицируются в качестве неклассифицированных линий. В настоящее время насчитывается в общей сложности 34 неклассифицированных линий HCV-6. В этом исследовании они были помечены от un1 до un34, чтобы отличить от 29 определенных подтипов. Следует отметить, что эта временная нумерация не предназначена для целей классификации, а только показывает генетическую сложность HCV-6. Учитывая, что многие другие еще не полностью секвенированы, общее количество потенциальных линий HCV-6 на уровне подтипа очень велико (Li et al., 2014a, Li et al., 2015). Следовательно, их формальное обозначение подтипа должно строго следовать рекомендациям, представленным в недавно расширенной номенклатурной системе HCV (Smith et al., 2014).
Важным замечанием в этом исследовании является то, что 96 изолятов HCV-6 были получены от иммигрантов из стран Азии, проживающих в Северной Америке. Как правило, такие штаммы не станут эпидемически-опасными, потому что они в основном циркулируют в конкретной общности иммигранов. Однако азиатские иммигранты часто участвуют в предоставлении потенциально-опасных услуг: татуирование, пирсинг, косметические манипуляции, включая маникюр, которые являются известными факторами риска вирусной парентеральной передачи. Хотя фактически расширение присутствие HCV-6 до сих пор не наблюдается, мы можем провести параллель с аналогичными ситуациями которые изначально наблюдались в качестве спорадических случаев в незначительном масштабе и в небольших клиниках, например то, что произошло с ВИЧ-1 и другими распространенными генотипами HCV, так как в обоих случаях они являются быстро развивающимися и распространяющимися вирусами и могут быть эффективно передаваться парентеральным путем (Ou et al., 1992, Esteban et al., 1996, Birch et al., 2000, Metzker et al., 2002, Bracho et al., 2005, González-Candelas et al., 2013 ).
В недавних исследованиях мы выявили большую вариативность экзогенных штаммов HCV у иммигрантов из разных стран мира живущих в Северной Америке (Li et al., 2009, Li et al., 2012, Li et al., 2013, Li et al., 2014a, Li et al., 2014b, Li et al., 2015; Lu et al., 2013, Lu et al., 2014b, Lu et al., 2015a). Принято считать, что в случае выполнения основных условий для передачи, любой из штаммов HCV может получить широкое распространение. Например, уникальные изоляты подтипа 3i, которые мы выделили среди ряда выходцев из кавказа в Квебеке, могут представлять собой штамм HCV, который проник в Северную Америку благодаря парентеральному пути передачи в сообществе иммигрантов (Murphy et al. , 2007, Li et al., 2014b). Не так давно мы исследовали вспышку HCV в сообществе на юге Китая, которая затронула сотни людей. Эта вспышка была вызвана главным образом штаммом подтипа 6a, который получил широкое распространение в связи с оказанием небезопасных медицинских услуг в небольшой клинике, из-за нарушения норм дезинфекции и стерилизации путем повторного использования игл и шприцев (Lu et al., 2015b). Эта вспышка HCV-6 указывает на то, что в субоптимальных санитарных условиях необычный штамм HCV, который потенциально, имеет свои особенности в терапии, может периодически передаваться в местном сообществе. Таким образом, анализ штаммов HCV-6 среди иммигрантов, и изучение его особенностей, не только дает информации для проведения будущих исследований, но и позволяет оперативно принимать изменения в мерах связанных с профилактикой и контролем HCV в Северной Америке.
Источник:
«Analysis of HCV-6 isolates among Asian-born immigrants in North America reveals their high genetic diversity and a new subtype»
Virology
Volume 492, May 2016 , Pages 25-31
https://doi.org/10.1016/j.virol.2016.01.028